 鯨鯊是大海中的明星生物 (余慧盈 攝)
鯨鯊是大海中的明星生物 (余慧盈 攝)
每年的六月八日為聯合國訂定的世界海洋日,由1992年在里約召開的第一屆地球高峰會中所選定,到了2008年聯合國正式訂立此日為官方紀念日,期能喚醒大眾對海洋環保與生態問題的重視。每年的世界海洋日都會有一個主題,例如2017與2018年的主題分別為「我們的海洋,我們的未來」(Our Oceans, Our Future)與「潔淨我們的海洋」(Preventing Plastic Pollution and Encouraging Solutions for a Healthy Ocean),2019年則延伸至近年熱門的性平議題,設定為「性別與海洋」(Gender and Oceans),提倡性別面向與海洋的關係。
由於人類對地球資源的需求日趨殷切,許多生態系遂面臨竭澤而漁與棲地破壞等前所未有的挑戰,科學家預測若人類對目前海洋生物的利用模式沒有改善的話,到2048年海洋將沒有可供常態性捕撈的經濟物種。除了過度捕撈的問題外,棲地開發及引入外來種的作為也嚴重威脅著海洋生態系。而且無論是陸域或海域,因人類而致的物種滅絕事件持續發生,由於規模太過廣泛了,因此被稱為是第六次大滅絕。雖然情勢看似絕望,所幸仍有一絲希望,因為在生物絕種持續發生的當下,有心人士對物種與棲地的保育觀念也隨著各種推廣活動方興未艾。
生物相調查是生態保育的基本功,對棲地的生物組成與物種分佈有初步認識後,才能針對特定物種或棲地類型推動適當的保育工作,例如台灣各地的國家公園就陸續進行著各類生物相調查,就是為生態保育建立基礎資料庫。水域方面,傳統調查方法可經由生物捕撈,如設陷阱、釣魚或拖網等方法捕撈生物上岸,以茲調查水生生物的分布及數量,或藉由潛水及浮潛直接觀察並計數。然而受限於動物的隱蔽性與複雜的棲地類型,生物相監測工作往往需要大量人力、物力及時間的投入,且可能具有相當的危險性,例如捕撈及潛水調查。
由於訓練外部形態進行物種鑑定的過程冗長,緩不濟急,若能有某種調查方法能取代或輔助傳統的方法,並降低調查的費用、減少對生態系統的干擾及降低採樣危險性,這對保育工作將是一大福音,例如近年來廣泛使用的分子技術,生命條碼(DNA barcoding)等即是,而且以這些方法來檢測生物相已日漸蓬勃發展。
自20世紀中DNA的結構被解密後,接續發展出DNA定序技術以及聚合酶連鎖反應,使其相關的研究蓬勃發展,應用面也越來越廣。但無論是那方面的應用,DNA多須從生物體的肌肉或血液等組織直接取得,甚為不便。其實生物排放的糞便、黏液、尿液與脫落的皮屑中也帶有DNA,若收集這些生物體釋放於環境中的組織或代謝物,經由特定程序處理後,仍能取得原物種之DNA,而這類從水、空氣、土壤等環境界質中取得的DNA就稱為環境DNA。這方法不需直接自生物組織提取DNA,故沒有生物採集的困擾,但藉由環境介質中殘留的DNA去比對資料庫中的序列,仍可進行辨認或偵測物種組成,此法已廣泛應用於水陸域的環境。不過技術或許可行,環境DNA的應用仍受限於DNA序列資料庫的豐富與否,對於資料庫中尚未收錄基因序列的物種與生物類群,環境DNA也無法發揮其作用。
環境DNA源自於微生物學,藉由沉積物中的生物碎屑,萃取其中的DNA並純化,將可自沉積物中獲得若干遺傳資訊。隨著技術的演進,環境DNA已應用於土壤、永凍土、化石和洞穴沉積岩等介質,生物類群囊括微生物至大型哺乳類,年代也可上溯永凍層到溫和環境中的古老生物相。早期的環境DNA定序工作均採用桑格定序法(Sanger sequencing),此方法係利用雙脫氧鏈終止定序法(dideoxy chain-termination),藉由聚合酶連鎖反應大量複製不同長短的目標DNA片段,再依據各長短DNA鏈最後一個鹼基的螢光訊號進行序列判讀。但因雙脫氧鏈終止定序法係針對相同長度的所有序列同時定序,若定序樣本中有兩種以上的序列,將造成機器判讀DNA訊號時互相干擾。受此限制,故桑格定序技術僅能針對單一DNA來源,而無法檢測複雜的生物組成或全面的生物相,也限制了環境DNA的應用。
惟隨著科技進展,近年有一次世代的新定序技術(Next generation sequencing, NGS)為環境DNA的利用開啟了嶄新的一頁。所謂次世代定序為桑格定序法以外的新一代定序技術的通稱,其差別在於次世代定序不是通過針對相同長度序列集體定序的雙脫氧鏈終止定序法,而是將序列打斷成差不多長度後,再針對每一條序列同時定序,因此沒有序列訊號彼此干擾的問題,因而可達到快速、大量、經濟且全面的定序。
 研究人員在東沙環礁進行調查工作 (魏儀 攝)
研究人員在東沙環礁進行調查工作 (魏儀 攝)
2010年Creer et al. 首次將高通量分子技術應用於環境DNA的研究,他利用次世代定序平台定序多個獨立樣本中生物的環境DNA訊號,分析海洋及熱帶雨林棲地,偵測多種植物及動物。結果顯示透過次世代定序技術可以有效的建立小型生物及複雜的真核生物群落的環境DNA資訊,為環境調查提供了相當的優勢。環境DNA也可應用於水生系統中,如Martellini et al. (2005)採集了河水樣本,檢測牛、豬和羊等牲畜的DNA,以茲控管家庭汙水排放及河流品質;另Minamoto et al. (2011)也透過採集河流水樣,調查及建立當地的淡水魚種組成。Thomsen et al. (2012b)則利用次世代定序,首次應用環境DNA來檢測海水魚多樣性及組成。和傳統調查法比較,顯示大部分經由傳統捕捉法紀錄的物種皆可被環境DNA偵測到,顯示環境DNA潛在的發展空間及物種調查能力良好。
然而,環境因子會影響環境DNA的降解速率,因而使人錯估了目標物種的族群量大小,如Takahara et al. (2012)自水族缸實驗中測得目標生物移除環境後,環境DNA序列數(copy number)會在六到九天內開始變化,且目標生物的活動性及族群量大小亦是影響環境DNA序列數的重要因素,但水溫則對於序列數沒有顯著性的影響。此外,Thomsen et al. (2012a)基於前人的研究成果,發現可測得許多物種的環境DNA會於生物移除後的兩週內降解至無法偵測,表示環境DNA只能偵測當下環境中的物種,但不會受到過去生物DNA累積的影響,更證實環境DNA偵測目標物種的可靠性。
之後,Yamamoto et al. (2016)針對魚群分布和生物量進行了實驗,他結合聲納技術後發現環境DNA的濃度和族群量有高度的正相關,可作為反映魚群生物量的重要指標。經前述各研究的比較後,發現環境DNA能偵測到傳統調查所得的63-100%的物種組成,並且能清楚觀測調查樣點間族群組成的差異性(Yamamoto et al. , 2017),顯示目前環境DNA調查野外族群量、組成及分布,已具有相當的參考價值。這些研究為環境DNA應用於監測野外族群量的時空分布及調查的準確性奠下了重要的基石,也顯示環境DNA的效率和即時性。然而儘管科技日新月異,目前仍無法建立環境DNA濃度和野外族群量的直接相關性。
環境DNA除了可應用於生物相的調查外,也可用以進行海洋瀕危物種的生態研究,藉由過濾海水樣本取代捕捉活體採取肌肉或血液組織,以避免對敏感生物的直接干擾。鯨鯊(Rhincodon typus)為廣受世人注目的海洋軟骨魚類,然而受到過度捕撈的影響,族群正瀕臨絕種的威脅,故成為野生動植物國際貿易公約(Convention on International Trade in Endangered Species of Wild Fauna and Flora, CITES)附錄II的瀕危動物。儘管它的體型較一般魚類龐大,但因採樣與觀察的困難,我們對於鯨鯊的生物資訊仍相當匱乏。若考慮經濟效益且不干擾鯨鯊的前提,僅需一桶水就能提供許多遺傳資訊的環境DNA此時就能派上用場了,提供方便又快速的研究管道。
Sigsgaard (2016) 採集了阿拉伯灣週遭海域鯨鯊的環境DNA,與現有的鯨鯊粒線體遺傳資訊比較後發現,環境DNA偵測到的遺傳訊息較傳統紀錄還多,顯示傳統經由收集鯨鯊組織而取得遺傳資訊的方式,不但耗時耗力,收效又有限。另外透過統計分析後也發現,阿拉伯灣的鯨鯊族群會和印度-太平洋地區的族群聚集,然而並不會和大西洋的族群聚集,也顯示了全球鯨鯊的遷徙與族群間互動的關係。鯊魚也是生物保育的焦點物種,為了避免傳統監測法需補撈個體而造成傷害,Bakker (2017) 透過環境DNA分析加勒比海及太平洋珊瑚礁海域的鯊魚種類及相對豐度,進而區分各地區的主要棲息種類。方便又經濟的環境DNA能夠有效地建構及調查大尺度的生物訊息,並補足傳統資訊的遺漏,強化生物監測及保育的效果。
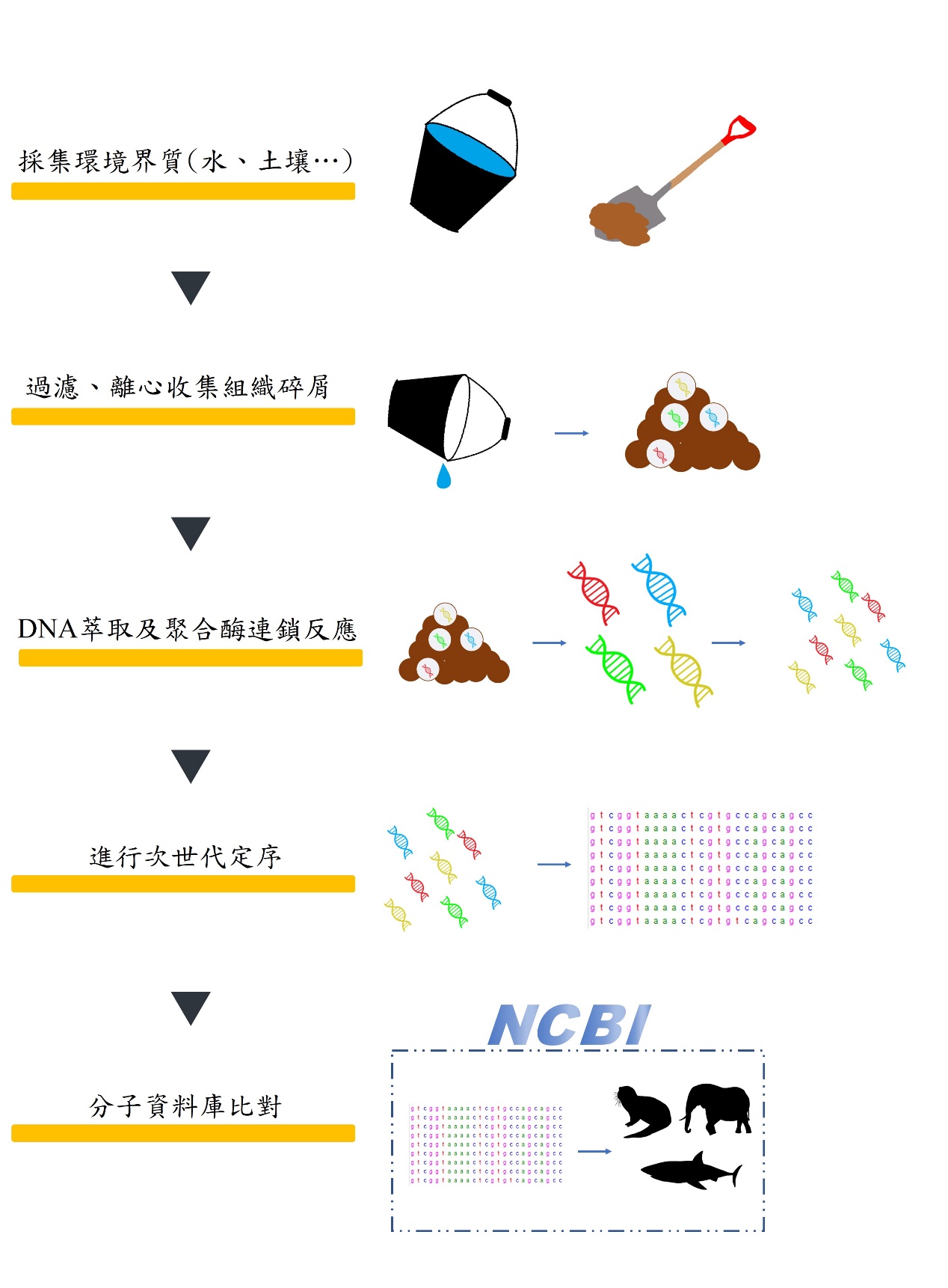 環境DNA採樣示意圖。於野外採集環境界質(如水、土壤等)過濾、離心後收集界質中的組織碎屑,透過DNA萃取及聚合酶連鎖反應放大目標序列片段後送往生技公司進行次世代定序,最終將序列上傳至分子資料庫辨識物種(劉祐辰 制)。
環境DNA採樣示意圖。於野外採集環境界質(如水、土壤等)過濾、離心後收集界質中的組織碎屑,透過DNA萃取及聚合酶連鎖反應放大目標序列片段後送往生技公司進行次世代定序,最終將序列上傳至分子資料庫辨識物種(劉祐辰 制)。
環境DNA也可應用於入侵種的偵測,取代傳統費時費力的採集觀察,以對應入侵種的快速擴張,期能有效地即時監測並盡早訂定遏止入侵種擴張的計畫。比如在歐洲,環境DNA就被利用來偵測馬珂蛤(Rangia cuneata)的擴散。馬珂蛤原生於墨西哥灣,透過壓艙水入侵歐洲,2005年首見於比利時,四年後即出現於波羅的海,且於2011年發現其最高密度每平方公尺可超過四千顆,嚴重威脅當地底棲生物相。為有效防止馬珂蛤的擴散,科學家開發快速了可檢測馬珂蛤環境DNA的技術,期能在牠們入侵初期就將之移除。儘管環境DNA能有效偵測外來種,但考慮海洋的開放水域特性,洋流的移動可能會影響環境DNA的分析結果,因此目前外來種檢測僅應用於淡水域。
在台灣,海洋國家公園管理處已將環境DNA應用於東沙環礁國家公園與澎湖南方四島國家公園的生態保育,做為補充魚類生物相與研究燕鷗食性的工具。魚類生物相研究主要係利用水樣分析以補充傳統潛水調查不易觀察的類群,並完善國家公園的魚類相基礎資料;燕鷗食性研究則是採集排遺樣本,分析其中的魚類組成以瞭解燕鷗攝食的魚類組成。環境DNA已被證實為是可靠且經濟快速的生態學研究工具,雖然其效力仍受限於資料庫的豐富程度與否,而且目前也無法做定量分析,但仍無損其方便性所帶來的效益,值得海洋保育等相關領域推廣應用。
參考資料
1.Takahara T, Minamoto T, Yamanaka H, Doi H, Kawabata Z (2012) Estimation of Fish Biomass Using Environmental DNA. PloS ONE 7 ((4)) e35868.
2.Thomsen PF, Kielgast J, Iversen LL et al. (2012a) Monitoring endangered freshwater biodiversity by environmental DNA. Molecular Ecology, 21, 2565–2573.
3.
Thomsen PF, J. Kielgast, L.L. Iversen, P.R. Møller, M. Rasmussen, E. Willerslev Detection of a diverse marine fish fauna using environmental DNA from seawater samples PLoS ONE, 7 (2012b), p. e41732, 10.1371/journal.pone.0041732.
 鯨鯊是大海中的明星生物 (余慧盈 攝)
鯨鯊是大海中的明星生物 (余慧盈 攝) 研究人員在東沙環礁進行調查工作 (魏儀 攝)
研究人員在東沙環礁進行調查工作 (魏儀 攝) 環境DNA採樣示意圖。於野外採集環境界質(如水、土壤等)過濾、離心後收集界質中的組織碎屑,透過DNA萃取及聚合酶連鎖反應放大目標序列片段後送往生技公司進行次世代定序,最終將序列上傳至分子資料庫辨識物種(劉祐辰 制)。
環境DNA採樣示意圖。於野外採集環境界質(如水、土壤等)過濾、離心後收集界質中的組織碎屑,透過DNA萃取及聚合酶連鎖反應放大目標序列片段後送往生技公司進行次世代定序,最終將序列上傳至分子資料庫辨識物種(劉祐辰 制)。