人工智慧與分子生物的距離是八竿子打不著?
你可能知道人工智慧可以幫助我們分析圖像,例如人臉辨識就是很耳熟能詳的一個應用,而背後的分析技術就是深度學習。而分子生物學是從分子層級來了解生物的一種科學,這跟一般生物學從比較巨觀的角度來了解生物有點不同。圖一說明了不同層級的生物學:觀察樹葉是綠色的,這是最巨觀的層級;進一步在顯微鏡中觀察樹葉的細胞、葉綠體,這是細胞層級;再進一步了解樹葉細胞內的葉綠素,才是分子層級。那麼,人工智慧和分子生物學之間有什麼關係呢?簡單的說,這篇文章要帶我們來看:如何把生物分子視為圖像,並利用類似人臉辨識的技術來進行分析。
圖一:不同層級的生物學,由左至右為巨觀層級的樹葉、細胞層級的葉綠體、分子層級的葉綠素。圖/Krzysztof P. Jasiutowicz、Mariana Ruiz LadyofHats、Jynto
深度學習跟分子生物學的範圍都很大,這篇文章會專注在將卷積神經網路(Coevolutionary Neural Network, CNN) 應用在辨識信號肽 (signal peptide)的問題上。
你要蓋出什麼樣的房子?深度學習與卷積神經網路
如圖二所示,人工智慧模型就像一棟房子,房子的採光、有沒有冬暖夏涼,反應了這個模型的好壞;而深度學習,或是說神經網路,則是一種蓋房子的「工法」。使用同樣工法的房子會有某些共通的特性,但是像一層的坪數、樓層的總數等等,還是可以因應需求而有所不同。神經網路可以說是近年來最熱門的工法,使用這種工法搭建的模型,使用起來效果特別好;它的特色是模型是一層一層建構的,每一層的結構都很簡單,從房子來說是可以蓋很高,從模型來說就是可以很輕易的加深。當神經網路模型深度漸漸大於其它工法搭建的模型時,才出現深度學習這個名詞,所以深度學習真的只是用比較「深」的模型來分析資料,照字面去理解完全沒有問題。
那卷積神經網路跟深度學習的關係又是什麼呢?它就是有用到卷積層 (convolutional layer) 的神經網路。卷積層聽起來有點饒口,它的命名其實來自於數學上的卷積運算,這種運算套用在圖像上時,效果是將鄰近的像素資料彙整起來,形成比單一像素本身更豐富的資訊。以人臉辨識來說,如果一次只看一個像素,大概很難辨識出什麼;而一次看整張圖片時,不是做不到,但有時比較難抓到規律。以人類來說,當人臉出現在圖片某個區域時,視野會自然聚焦在這個區域,並忽略其它區域的資訊。因此,卷積層這種整合鄰近區域資料的能力,正好符合人眼的特性,在圖像辨識上就能發揮很好的效果。
整體來看,神經網路是講整個模型的工法,規定了層與層之間怎麼接起來,而卷積層是單指某一層的工法。卷積神經網路通常是講整個模型中,至少有一層是卷積層。那如果全部層都用卷積層可以嗎?當然可以,你可以稱它為全卷積網路 (Fully Convolutional Network, FCN),這篇文章介紹的深度學習模型就屬於這種神經網路。
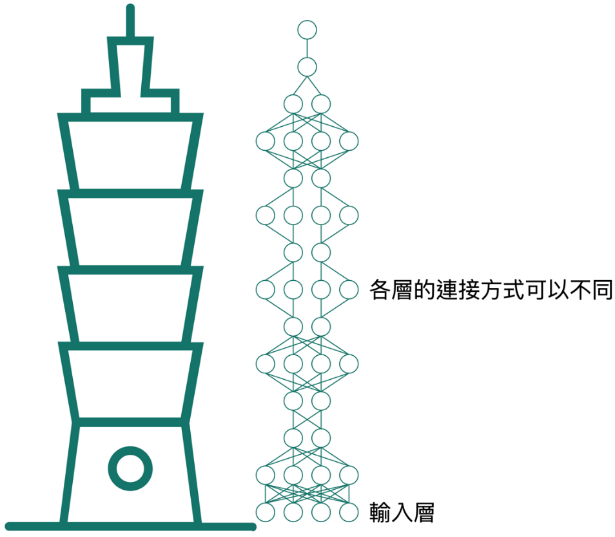
圖二:深度學習模型就像房子般一層一層蓋起來,每一層的「工法」可以不同,可以依需求彈性調整模型架構。圖/Taipei by mikicon from the Noun Project
「信號肽」是什麼?
就像植物有葉綠素一樣,人體內也有很多「素」,例如血紅素、胰島素、腎上腺素等等,這些素都是在細胞內很重要的一種生物分子:「蛋白質」,生物的運作就是靠各式各樣的蛋白質合作才能維持。整個蛋白質工作網路是一個龐大且複雜的系統,蛋白質在發揮其功能前,需要先被製造並運送至細胞內正確的位置,而信號肽是蛋白質前端的一小部分,在運送過程中扮演了重要的角色,用來指導蛋白質跨膜轉移,也稱作訊號序列。準確辨識信號肽能夠幫助我們進一步了解蛋白質運送的過程,這篇文章介紹的方法將蛋白質當作一張圖像,試圖找出信號肽的規律,未來就可以用這些規律辨識蛋白質是否帶有信號肽,就像辨識圖像中是否有人臉一樣。
運用資訊、計算技術幫助生物研究稱為生物資訊 (bioinformatics) 或是計算生物學 (computational biology),其中序列分析指的是分析生物序列的特徵、功能、結構以及演化,辨識信號肽又只是其中一種應用。目前估計人體內大約有八萬到四十萬種蛋白質,加上其他物種,蛋白質相關的知識還有很多研究空間,但也需要大量的資源投入。相較於其它技術,序列分析的特色是「快」,它的角色有點像是快篩,快速過濾大量不可能的狀況,讓後面的資源能夠投入到比較有機會成功的地方。而現在快速定序技術越來越成熟,序列資料正以指數在成長,這個快篩的角色也會越來越重要。
要怎麼將圖像辨識的技術應用在辨識信號肽的問題上—U-Net
醫學影像比起一般的圖像,例如人臉辨識來說,資料取得的難度比較高。U-Net 是 Ronneberger 等幾位研究者在 2015 年提出的神經網路,它可以處理資料較少的應用,並在醫學影像辨識上取得了很好的效果。圖三是 U-Net 的架構,中間每個箭頭就是神經網路的一層,矩形則是該層輸出的結果,上面的數字表示一個像素帶有多少維度的資訊。舉例來說,它的輸入是 572x572 的灰階圖像,每個灰階圖像的像素可以用 0~255 的純量表示該像素有多「亮」,0 代表黑色,255 代表白色。這邊將純量視為維度為 1 的向量,所以輸入層上面的數字為 1,整張圖片可以視為 572x572x1 的矩陣。經過第一層之後,每個像素會變成 64 維的向量,最後輸出每個像素會用一個 2 維向量來表示該像素是否帶有要辨識的物件。U-Net 裡每一層根據工法的不同,可以分成四類,圖上以不同顏色的箭頭表示,其中藍色箭頭表示卷積層。往下的箭頭對圖像來說有縮小的效果,例如一張 572x572 的圖像,在通過一次往下的箭頭後會變成 284x284 的圖像;反之,往上的箭頭對圖像來說有放大的效果,整個 U-Net 有點像先將圖像縮小後,再慢慢放大回來。請想像你緊貼著一面巨大的牆,在這樣的狀況下要看清牆上畫了什麼圖是很困難的,但若是往後退個幾步,要看清牆上圖像的全貌就不是難事了。這個往後退的動作,就像是縮小圖像,也就是 U-Net 中往下的箭頭;當你知道整張圖的重點大概在哪邊,想進一步看細節時,可以再往前進,對應的就是 U-Net 中往上的箭頭(放大)。這個架構圖本身像英文字母 U,也是它被稱為 U-Net 的原因。
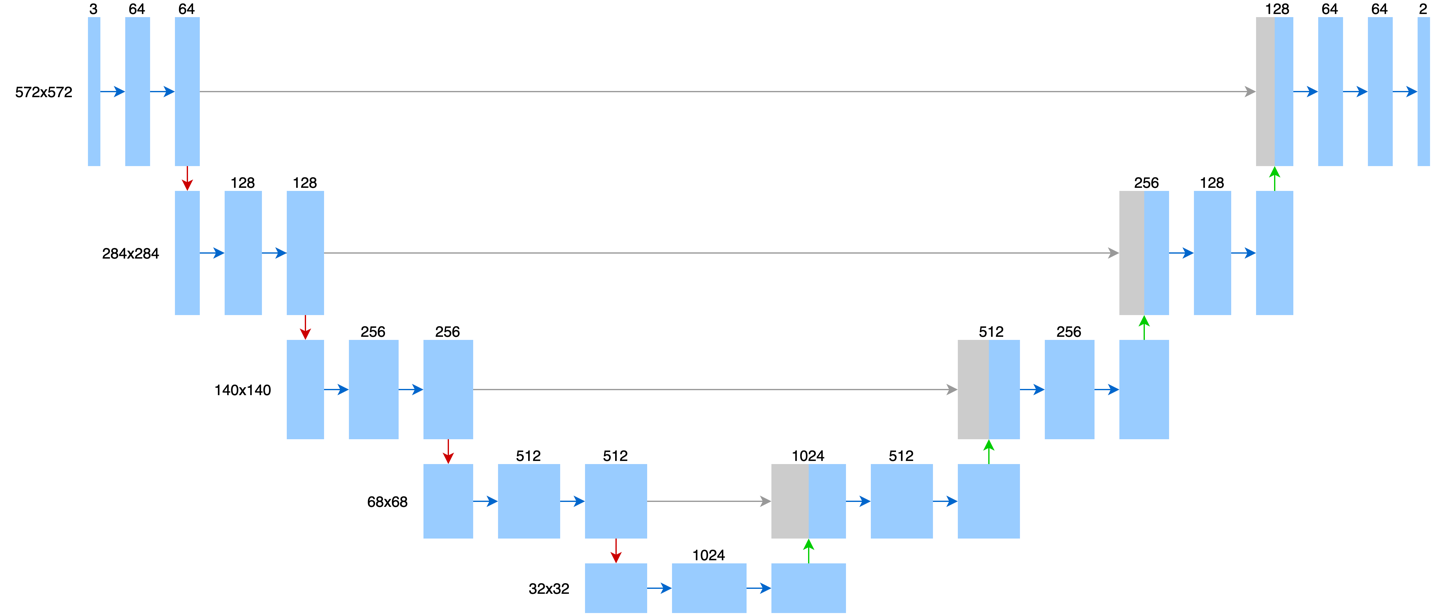
圖三:U-Net 的網路架構(藍色箭頭表示卷積層)
U-Net 的升級版—為分析蛋白質序列量身訂做的 SigUNet
在生物體內,將蛋白質運送到其功能發揮部位的過程稱為蛋白質分選 (protein sorting) ,在這個過程中,可以把信號肽想成火車的車票,它確保蛋白質可以搭乘大眾運輸工具(內質網 (endoplasmic reticulum))。信號肽這個車票很有趣的一點是,它是直接連在蛋白質上的,可以想成剪票後的票根就是蛋白質本身,而被剪掉的部分就是信號肽,它在上車後就不需要了。這種票根就是本體的設計有個問題,蛋白質是一種化學物質,它不像一般車票上會有明確分隔的虛線,讓人一目瞭然知道票根的位置在哪裡。請把蛋白質想成一個長鏈狀、一環一環串在一起的分子,每一個環就是一顆胺基酸 (amino acid),而信號肽是蛋白質前端大約 11 到 27 顆胺基酸。再進一步細看信號肽本身的話,可以大致分成三個區段,最前面是長度大約 1 到 5 的正電區段,也就是這說區段的胺基酸大都帶正電荷;接著是長度大約 7 到 15 的疏水區段;以及最後長度大約 3 到 7 的極性區段。
一般計算分析人員拿到的蛋白質長得會像一個英文字母序列(如圖四所示),每個字母代表一個胺基酸,例如 G 代表甘胺酸 (Glycine)、A 代表丙胺酸 (Alanine)。整個辨識信號肽的問題,就會變成是輸入一串英文字母序列,然後辨識每個字母是否為位於信號肽區段。這篇文章介紹的辨識信號肽模型實際使用的是 U-Net 改良版—SigUNet(架構如圖五所示)。SigUNet 將這個英文字母序列視為一張圖像,一個字母視為圖像上的一個像素,並根據這樣的假設來調整 U-Net。怎麼進行調整呢?
整體來說,SigUNet 的輸入變為 96x20,其中 96 是序列的長度,對應到 U-Net 的 572x572x1 中的 572x572;而 x20 對應到 U-Net 的 x1。此外,由於信號肽的資料量又比醫學影像更少,所以 SigUNet 的網路複雜度有進一步簡化。圖中可以看到每層的維度都有所下降,在 U-Net 中,當圖像縮到最小時,單一像素會用高達 1024 維的向量來表示;而在 SigUNet 中,當序列縮到最短時,單一字母最高也只需要使用 40 維的向量來表示。此外,U-Net 經過四次縮小以及四次放大,而 SigUNet 只經過三次縮小以及三次放大。
圖四:信號肽的結構與編碼。
計算分析人員拿到的蛋白質會是一個英文字母序列,每個字母代表一種胺基酸,如圖中的 MRRGR…。序列前端信號肰的部分可以區分為正電區段、疏水區段以及極性區段。每個字母會用一個 20 維的 0/1 向量來表示,以長度 96 的序列為例,對深度學習模型來說就是一個 96x20 的矩陣。注意圖中正電區段、疏水區段以及極性區段這樣的資訊並不會輸入模型,相反的,模型要從這 96x20 的矩陣中辨識這些區段,並依此預測輸入的英文字母序列是否帶有信號肽。
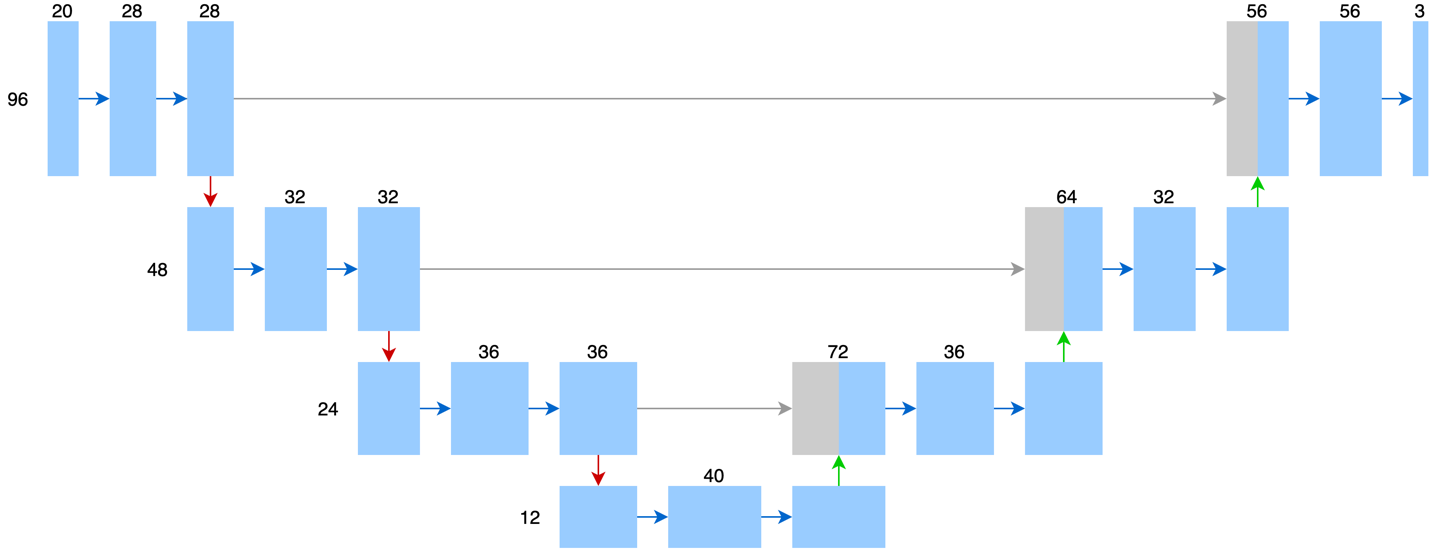
圖五:SigUNet的網路架構。
上面這些調整使得 SigUNet 在辨識信號肽的表現比 U-Net 好上許多,不過這個例子主要是想讓大家了解,原來圖像深度學習網路也可以這樣運用。如果拋開改動的細節,SigUNet 完全保留了 U-Net 的精神—先退後看全貌再前進看細節。
蛋白質序列對許多人來說與圖像不同,是非常陌生的東西,但實驗結果顯示出蛋白質序列也可以採用同樣的分析邏輯。希望大家讀到這裡,會覺得生醫領域的應用也不是那麼可怕,並且可以開始想像,說不定還有許多圖像分析技術可以運用在分子生物學上;或是反過來想,這篇文章提到的分析技術,有沒有機會套用分子生物學以外的領域呢?總之,下次觸及不同領域時,或許不用太害怕突如其來的專有名詞,下一點工夫去理解,反而可以把它們想成機會,好好享受一下跨領域的挑戰與樂趣!
本著作係採用 創用 CC 姓名標示─非商業性─禁止改作 3.0 台灣 授權條款 授權.
本授權條款允許使用者重製、散布、傳輸著作,但不得為商業目的之使用,亦不得修改該著作。 使用時必須按照著作人指定的方式表彰其姓名。
閱讀授權標章或
授權條款法律文字。